4.1 缺失数据的处理
在实际应用过程中,样本由于各种原因缺少一个或多个值的情况并不少见。其原因主要有:数据采集过程中出现了错误,常用的度量方法不适用于某些特征,或者在调查过程中某些数据未被填写,等等。通常,我们见到的缺失值是数据表中的空值,或者是类似于NaN(Not A Number,非数字)的占位符。
遗憾的是,如果我们忽略这些缺失值,将导致大部分的计算工具无法对原始数据进行处理,或者得到某些不可预知的结果。因此,在做更深入的分析之前,必须对这些缺失值进行处理。而在讨论处理缺失值的几个技巧之前,我们先通过一个CSV(comma-separed values,以逗号为分隔符的数值)文件构造一个简单的例子,以便更好地把握问题的核心所在:
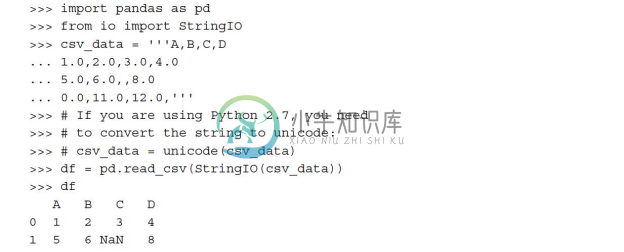
![]()
在上述代码中,我们通过read_csv函数将CSV格式的数据读取到pandas的数据框(DataFrame)中,可以看到:其中有两个缺失值由NaN替代。示例代码中的StringIO函数在此仅起到演示作用:如果我们的数据是存储在硬盘上的CSV文件,就可以通过此函数以字符串的方式从文件中读取数据,并将其转换成DataFrame的格式赋值给csv_data。
对于大的DataFrame来说,手工搜索缺失值是极其繁琐的,在此情况下,我们可以使用代码中的isnull方法返回一个布尔型的DataFrame值,若DataFrame的元素单元包含数字型数值则返回值为假(False),若数据值缺失则返回值为真(True)。通过sum方法,我们可以得到如下所示的每列中缺失值数量:

通过这种方式,我们可以得到每列中缺失值的数量。在下面的小节中,我们将学习几种不同的处理缺失数据的策略。
 虽然scikit-learn是在NumPy数组的基础上开发的,但有时使用pandas的DataFrame来处理数据会更为方便。在我们使用scikit-learn处理数据之前,可以通过DataFrame的value属性来访问相关的NumPy数组:
虽然scikit-learn是在NumPy数组的基础上开发的,但有时使用pandas的DataFrame来处理数据会更为方便。在我们使用scikit-learn处理数据之前,可以通过DataFrame的value属性来访问相关的NumPy数组:

4.1.1 将存在缺失值的特征或样本删除
处理缺失数据最简单的方法就是:将包含确实数据的特征(列)或者样本(行)从数据集中删除。可通过dropna方法来删除数据集中包含缺失值的行:

类似地,我们可以将axis参数设为1,以删除数据集中至少包含一个NaN值的列:

dropna方法还支持其他参数,以应对各种缺失值的情况:

![]()
删除缺失数据看起来像是一种非常方便的方法,但也有一定的缺点,如:我们可能会删除过多的样本,导致分析结果可靠性不高。从另一方面讲,如果删除了过多的特征列,有可能会面临丢失有价值信息的风险,而这些信息是分类器用来区分类别所必需的。在下一节,我们将学习另外一种最常用的处理缺失数据的方法:插值技术。
4.1.2 缺失数据填充
通常情况下,删除样本或者删除数据集中的整个特征列是不可行的,因为这样可能会丢失过多有价值的数据。在此情况下,我们可以使用不同的插值技术,通过数据集中其他训练样本的数据来估计缺失值。最常用的插值技术之一就是均值插补(meaneinputation),即使用相应的特征均值来替换缺失值。我们可使用scikit-learn中的Impute类方便地实现此方法,代码如下:

在此,首先计算各特征列的均值,然后使用相应的特征均值对NaN进行替换。如果我们把参数axis=0改为axis=1,则用每行的均值来进行相应的替换。参数strategy的可选项还有median或者most_frequent,后者代表使用对应行或列中出现频次最高的值来替换缺失值,常用于填充类别特征值。
4.1.3 理解scikit-learn预估器的API
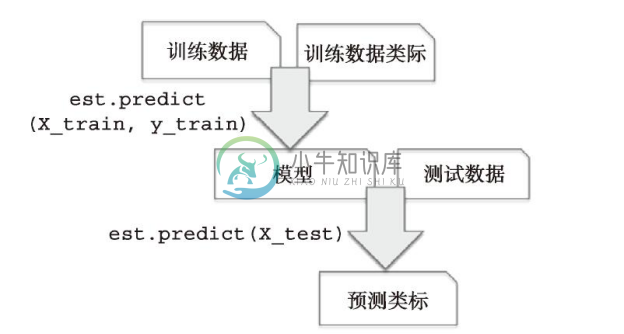
在上一节中,我们使用了scikit-learn中的Imputer类来填充我们数据集中的缺失值。Imputer类属于sciait-learn中的转换器类,主要用于数据的转换。这些类中常用的两个方法是fit和transform。其中,fit方法用于对数据集中的参数进行识别并构建相应的数据补齐模型,而transform方法则使用刚构建的数据补齐模型对数据集中相应参数的缺失值进行补齐。所有待补齐数据的维度应该与数据补齐模型中其他数据的维度相同。下图解释了转换器类如何对训练和测试数据进行数据处理(在此是数据补齐):

我们在第3章中用到了分类器,它们在scikit-learn中属于预估器类别,其API的设计与转换器类非常相似。在后续内容中我们将看到,预估器类包含一个predict方法,同时也包含一个transform方法。读者应该记得,我们在训练预估器用于分类任务时,同样使用了一个fit方法来对参数进行设定。在监督学习中,我们额外提供了类标用于构建模型,而模型可通过predict方法对新的样本数据展开预测,如下图所示: