如何给sns.clustermap一个预先计算的距离矩阵?
通常当我做树状图和热图时,我使用距离矩阵并做很多SciPy事情。我想尝试一下,Seaborn但是Seaborn想要我的数据是矩形的(行=样本,cols
=属性,而不是距离矩阵)?
我本质上想seaborn用作后端来计算我的树状图并将其附加到我的热图上。这可能吗?如果没有,将来是否可以提供此功能。
也许我可以调整一些参数,以便可以使用距离矩阵而不是矩形矩阵?
这是用法:
seaborn.clustermap¶
seaborn.clustermap(data, pivot_kws=None, method='average', metric='euclidean',
z_score=None, standard_scale=None, figsize=None, cbar_kws=None, row_cluster=True,
col_cluster=True, row_linkage=None, col_linkage=None, row_colors=None,
col_colors=None, mask=None, **kwargs)
我的代码如下:
from sklearn.datasets import load_iris
iris = load_iris()
X, y = iris.data, iris.target
DF = pd.DataFrame(X, index = ["iris_%d" % (i) for i in range(X.shape[0])], columns = iris.feature_names)
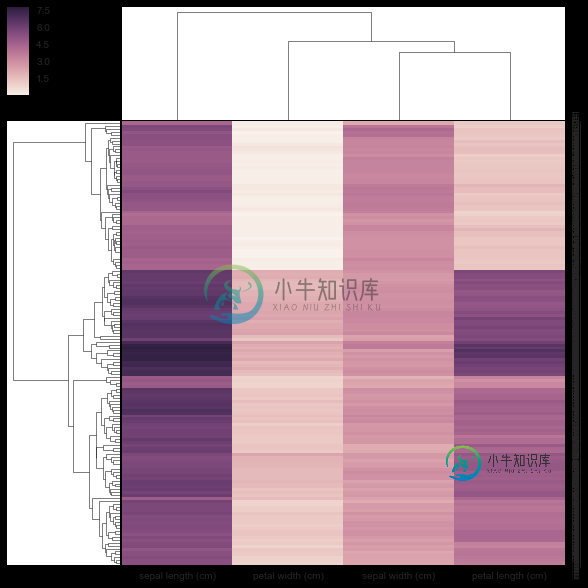
我认为下面的方法不正确,因为我给了它一个预先计算的距离矩阵,而不是它要求的矩形数据矩阵。没有关于如何使用相关性/距离矩阵的示例,clustermap但是有https://stanford.edu/~mwaskom/software/seaborn/examples/network_correlations.html的示例,但是排序不是与普通sns.heatmap函数一起进行的。
DF_corr = DF.T.corr()
DF_dism = 1 - DF_corr
sns.clustermap(DF_dism)

问题答案:
您可以将预先计算的距离矩阵作为链接传递给clustermap():
import pandas as pd, seaborn as sns
import scipy.spatial as sp, scipy.cluster.hierarchy as hc
from sklearn.datasets import load_iris
sns.set(font="monospace")
iris = load_iris()
X, y = iris.data, iris.target
DF = pd.DataFrame(X, index = ["iris_%d" % (i) for i in range(X.shape[0])], columns = iris.feature_names)
DF_corr = DF.T.corr()
DF_dism = 1 - DF_corr # distance matrix
linkage = hc.linkage(sp.distance.squareform(DF_dism), method='average')
sns.clustermap(DF_dism, row_linkage=linkage, col_linkage=linkage)
对于clustermap(distance_matrix)(即,未通过链接),链接是基于距离矩阵中行和列的成对距离(在内部获得详细信息,在内部进行计算),而不是直接使用距离矩阵的元素(正确的解决方案)
。结果,输出与问题中的输出有些不同:
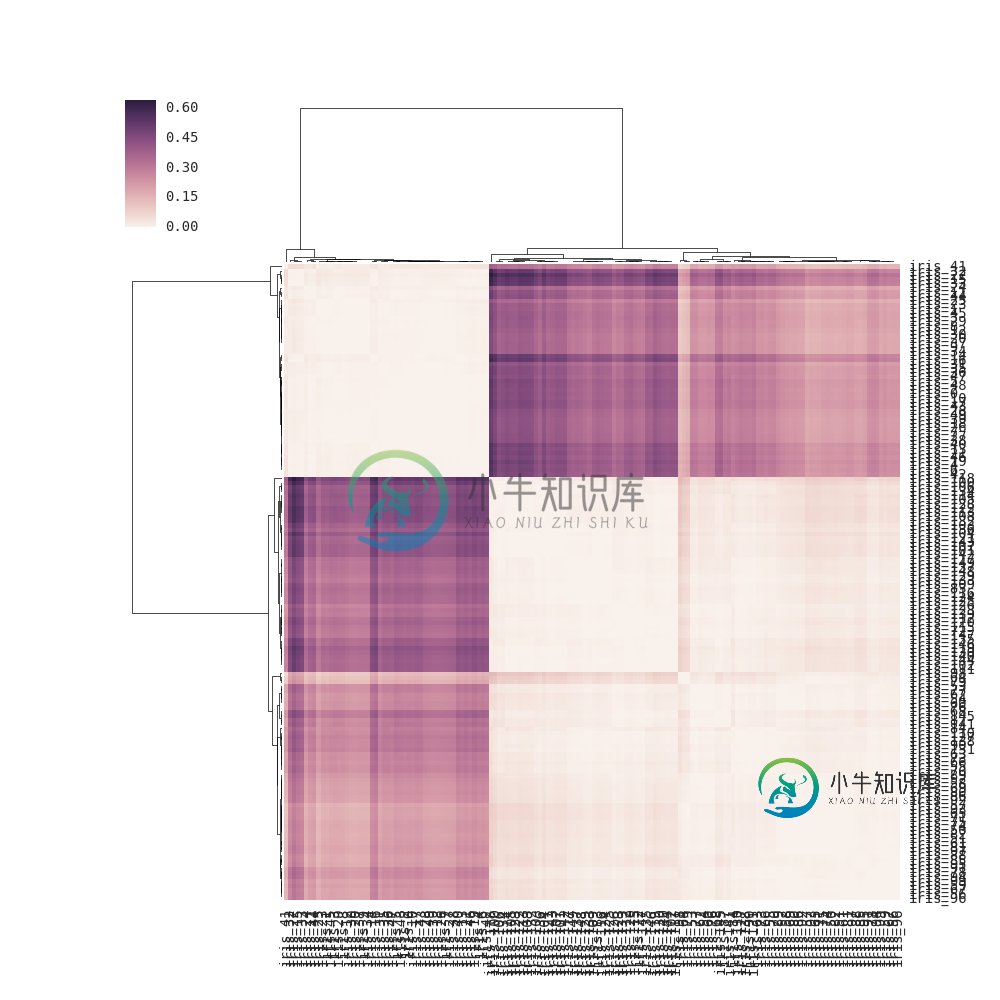
注意:如果没有row_linkage将传递给clustermap(),则通过将每行视为一个“点”(观察)并计算点之间的成对距离来内部确定行链接。因此行树状图反映了行相似性。与相似col_linkage,其中每列均视为一个点。此说明可能应该添加到docs中。在此修改文档的第一个示例,以使内部链接计算明确:
import seaborn as sns; sns.set()
import scipy.spatial as sp, scipy.cluster.hierarchy as hc
flights = sns.load_dataset("flights")
flights = flights.pivot("month", "year", "passengers")
row_linkage, col_linkage = (hc.linkage(sp.distance.pdist(x), method='average')
for x in (flights.values, flights.values.T))
g = sns.clustermap(flights, row_linkage=row_linkage, col_linkage=col_linkage)
# note: this produces the same plot as "sns.clustermap(flights)", where
# clustermap() calculates the row and column linkages internally
-
我想用Optaplanner解决VRP问题(添加一些内容)。在文档中,人们经常说,预先计算位置之间的距离,然后对每个位置使用
-
问题内容: 为了在地图上画一个圆,我有一个中心GLatLng(A)和一个半径(r)以米为单位。 这是一个图: 如何计算位置B的GLatLng?假设r平行于赤道。 使用GLatLng.distanceFrom()方法获得给定A和B时的半径是微不足道的-但反之则不然。似乎我需要做一些更重的数学运算。 问题答案: 我们将需要一种方法,该方法会在给定方位角和距源点的距离时返回目标点。幸运的是,克里斯·韦尼
-
我需要计算存储在csr稀疏矩阵和一些点列表中的所有点之间的欧氏距离。对我来说,将csr转换为稠密的csr会更容易,但由于内存不足,我无法将其转换为稠密的csr,因此我需要将其保留为csr。 例如,我有一个数据\u csr稀疏矩阵(csr和稠密视图): 这个中心点列表: 使用包,data_csr和中心之间的欧几里德距离数组将像下面这样。因此,在center的每行中,总共6个点中的每一个点都是根据da
-
虽然这段代码可以工作,但它本可以组织的更好。既然我们已经写了一个原型,那么我们就处于评价其设计并改进之的有利位置了。 那现在的代码有些什么问题呢? 我们提前不知道要创建多大的距离矩阵,所以我们选择了一个任意大的数字(50),然后创建了一个固定大小的矩阵。更好的方式是允许距离矩阵以类似Set的方式扩充,而apmatrix类的resize函数使之成为可能。 矩阵中的数据没有很好的封装。我们不得不以城市
-
我需要计算汽车行驶的距离!不是距离,不是距离到否。如果我们通过谷歌提供的API计算,距离可以完全不同。谷歌可以提供从一个点到另一个点的1公里距离,但汽车可以按照骑手想要的方式行驶800米。使用加速计没有帮助。它适用于步行,但绝不适用于更快的速度。 我尝试过使用Google的位置API:距离到或距离之间根本不是一个选项。它可以给出与IN REAL截然不同的结果。在真实的汽车中,可以通过非常短的地方并
-
最后,我们准备把数据从文件读入一个矩阵中。具体来说,每个城市在该矩阵中都一个相应的行和列。 我们将在main函数中创建该矩阵,它会剩余大量空间: apmatrix<int> distances (50, 50, 0); 在processLine内部,我们从Set中得到两个城市的索引,并以这两个索引为矩阵的索引,向矩阵中添加了新信息: int dist = convertToInt (distSt
-
我使用行人库(使用ps源、ping Goto和ps汇),想要模拟人行道环境。这个模型的目标是得到行人之间的距离小于1m的数据。所以,我尝试计算行人之间的距离。在Any逻辑中,可以通过使用getX()、getY和getId(可以每秒计算)来收集行人的信息。但是我不知道如何选择行人代理并计算它们之间的距离。我的意思是,如果有10个行人(id:1, 2, 3...),如何每秒钟获得1和2、1和3、2和3
-
我对计算两个numpy阵列(x和y)之间的各种空间距离感兴趣。 http://docs.scipy.org/doc/scipy-0.14.0/reference/generated/scipy.spatial.distance.cdist.html 但是,上述结果会产生太多不需要的结果。我怎样才能限制它只用于我所需的结果。 我想计算[1,11]和[31,41]之间的距离;[2,22]和[32,42

