OpenMM 是一个用于分子模拟的高性能工具包。可以将其用作库调用或作为独立程序运行。
此工具包还包含适用于 Python、C、C++ 与 Fortran 的语言绑定。
这一工具包具有极高的灵活性与速度,通过 GPU 加速以及 AMD、NVIDIA 和 Intel 集成 GPU 的优化实现了卓越的性能。
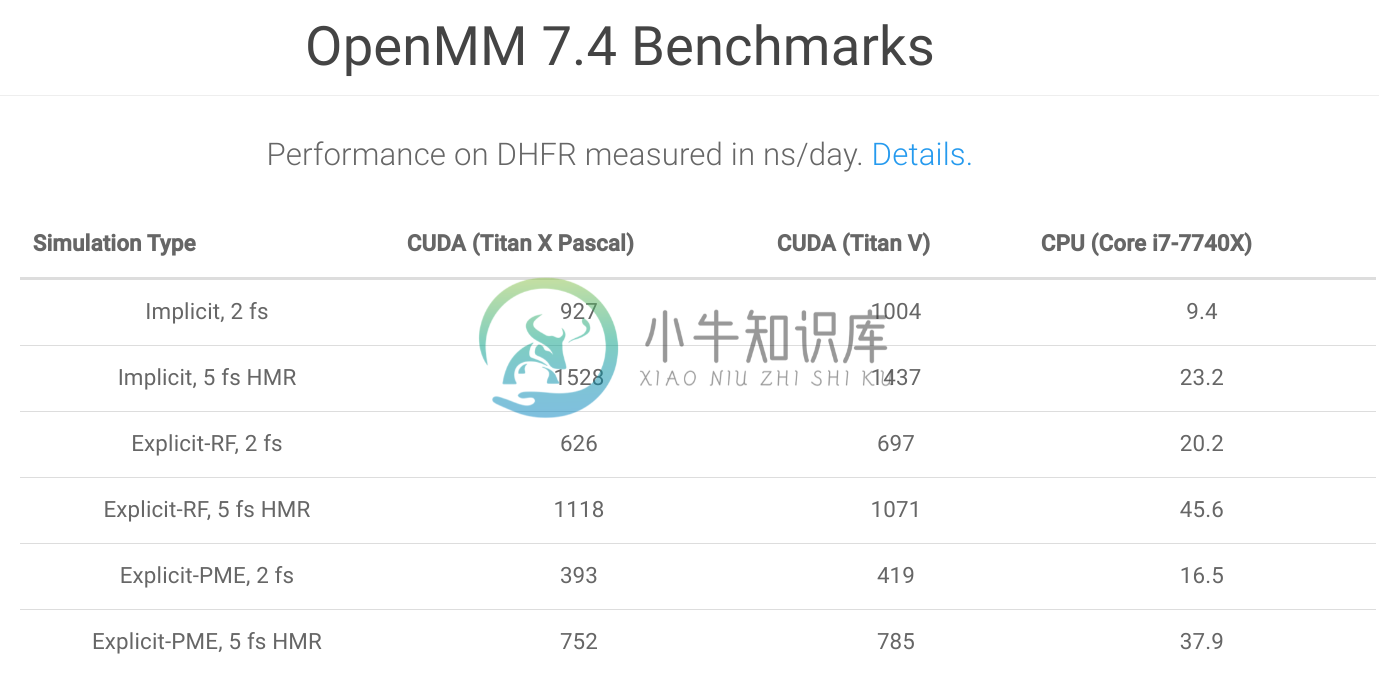
基准测试:
快速使用(Python):
from simtk.openmm.app import *
from simtk.openmm import *
from simtk.unit import *
from sys import stdout
pdb = PDBFile('input.pdb')
forcefield = ForceField('amber99sb.xml', 'tip3p.xml')
system = forcefield.createSystem(pdb.topology, nonbondedMethod=PME, nonbondedCutoff=1*nanometer, constraints=HBonds)
integrator = LangevinIntegrator(300*kelvin, 1/picosecond, 0.002*picoseconds)
simulation = Simulation(pdb.topology, system, integrator)
simulation.context.setPositions(pdb.positions)
simulation.minimizeEnergy()
simulation.reporters.append(PDBReporter('output.pdb', 1000))
simulation.reporters.append(StateDataReporter(stdout, 1000, step=True, potentialEnergy=True, temperature=True))
simulation.step(10000)-
报错原因 Alphafold2的官方目前只支持OpenMM==7.5.1, 但是目前OpenMM的最新版本已经更新到了7.7.0,如果你使用的是最新版本的OpenMM,则由于最新版本的OpenMM取消了simtk包名前缀,故导致了该问题,所以如果你想继续使用OpenMM==7.7.0,则你可能需要对alphafold2的代码进行部分的修改 解决原因 非Docker方式 # alphafold/al
-
1. 配置python3.6版本的anaconda3-5.2.0 从网址https://repo.continuum.io/archive/上寻找对应的版本 Anaconda3-5.2.0-Linux-x86.sh 507.3M 2018-05-30 13:05:46 81d5a1648e3aca4843f88ca3769c0830 用以下命令来下载文件到服务器 wget https://re
-
环境 Python=3.8 OpenMM=7.6 蛋白质的分子动力学轨迹分析 导入库 import MDAnalysis as mda from MDAnalysis.analysis import align, rms from MDAnalysis.tests.datafiles import PSF, DCD #These are testing files from MDanalysis,
-
环境 Python=3.8 OpenMM=7.6 PDBFixer 用于蛋白质预处理 导入库 import openmm as mm import openmm.app as app from openmm import unit import pdbfixer 载入数据 fixer = pdbfixer.PDBFixer('data/3poz.pdb') 设置力场 forcefield = ap
-
FFTW:是一个C子程序库,用于计算一个或多个维度,任意输入大小,实数和复数数据(以及偶数/奇数数据,即离散余弦/正弦变换)的离散傅里叶变换(DFT)或DCT.DST OpenMM:用于分子模拟的高性能工具包。将其作库或应用程序。用于测试性生物分子模拟的Omnia工具套件的一部分 PDBFixer:一个易于使用的应用程序,用于修复蛋白质数据库文件中的问题,为模拟他们做准备。他可以修复一下问题: (
-
OpenMMLab是一个适用于学术研究和工业应用的开源算法体系,涵盖了计算机视觉的许多研究课题,于2018年10月启动。主要包括2部分:开源项目和开放数据集。以下内容主要摘自于:https://openmmlab.com/ 开源项目:https://github.com/open-mmlab ,包含很多项目,由于每个项目起始开发时间不同,因此各个项目发布版本并不统一,有些项
-
从2018年年中开始,香港中文大学多媒体实验室(MMLab)就启动了OpenMMLab(https://github.com/open-mmlab)计划。这项计划的初衷是为计算机视觉的一些重要方向建立统一而开放的代码库,并不断把新的算法沉淀其中。我们相信,这一项工作可以推动可复现算法生态的建立,也是对计算机视觉社区的重要贡献。 主要项目包括: 目标检测-mmdetection, OpenMMLab
-
SAP MM 实施项目里Open PO 迁移思路探讨 .序言. SAP项目上线前夕,除了静态主数据需要导入以外,可能还有一些动态数据,比如open的采购订单,open的销售订单等单据也要迁移到SAP系统里。 笔者所在的项目里,对于Open PO 的迁移,是MM模块里一件非常重要的工作任务。笔者通过参与这个项目,了解到客户global template中Open PO的迁移策略,很受
-
问题内容: 我有2张桌子,和。用户可以有很多游戏。我需要所有有人数的人,以及他们的人数(有专栏的)。 附言:我需要将所有数据加载到管理表中。由于游戏太多。我决定对数据进行分页和限制。但是,甚至限制以下查询也需要花费相同的时间。如何更好地查询? 问题答案: 您可以在下面尝试使用表达式
-
本文向大家介绍为何说虚拟DOM会提高性能?相关面试题,主要包含被问及为何说虚拟DOM会提高性能?时的应答技巧和注意事项,需要的朋友参考一下 虚拟dom相当于在js和真实dom中间加了一个缓存,利用dom diff算法避免了没有必要的dom操作,从而提高性能
-
本文向大家介绍为什么虚拟dom会提高性能?相关面试题,主要包含被问及为什么虚拟dom会提高性能?时的应答技巧和注意事项,需要的朋友参考一下 虚拟dom相当于在js和真实dom中间加了一个缓存,利用dom diff算法避免了没有必要的dom操作,从而提髙性能。 具体实现步骤如下: 1. 用JavaScript对象结构表示DOM树的结构;然后用这个树构建一个真正的DOM树,插到文档当中 2.当状态变更
-
问题内容: 我在Python中使用时遇到了一些困难: 测试实际上返回正确的值,但它是Mock对象,不是。您如何在Python库中模拟属性? 问题答案: 您需要使用和: 这意味着:调用时,在该调用的返回值上,为属性设置a以返回value 。
-
我需要一些关于如何使用ScalaMock在类内模拟高阶函数的帮助 正如您在上面的代码中所看到的,当您传入一个具有高阶函数的值时,被模拟的函数可以正常工作,但如果您在每个位置键入它,则不会正常工作。在我的用例中,我不能像在第二次测试中那样做 下面是关于用例的更多信息,但对回答这个问题并不完全必要 这是一个简化的示例,但我需要一种方法使前者工作。原因是(我会尽我最大的努力解释这一点)我有一个正在测试的
-
我正在对二氧化硅进行分子动力学模拟。一段时间前,我转向波动偶极子模型,经过很大的努力,我仍然有问题实现它。 简而言之,系统中的所有氧原子都是可极化的,它们的偶极矩取决于它们相对于系统中所有其他原子的位置。更具体地说,我使用TS势(http://digitallibrary.sissa.it/bitstream/handle/1963/2874/tangney.pdf?sequence=2),其中偶

