Spark Java PCA:用于洗牌的Java堆空间和丢失的输出位置
我试图对一个包含4.827行和40.107列的dataframe进行PCA,但我接受了一个Java堆空间错误和丢失的输出位置进行洗牌(根据executors上的sdterr文件)。错误发生在PCA的“rowmatrix.scala:122”阶段。
集群
它是一个独立的集群,有16个工作节点,每个工作节点有一个执行器,有4个核心和21.504MB内存。主节点有15G内存,我用“java-jar-xmx15g myapp.jar”给它。另外,“spark.sql.shuffle.partitions”是192,“spark.driver.maxresultsize”是6g。
df1.persist (From the Storage Tab in spark UI it says it is 3Gb)
df2=df1.groupby(col1).pivot(col2).mean(col3) (This is a df with 4.827 columns and 40.107 rows)
df2.collectFirstColumnAsList
df3=df1.groupby(col2).pivot(col1).mean(col3) (This is a df with 40.107 columns and 4.827 rows)
-----it hangs here for around 1.5 hours creating metadata for upcoming dataframe-----
df4 = (..Imputer or na.fill on df3..)
df5 = (..VectorAssembler on df4..)
(..PCA on df5 with error Missing output location for shuffle..)
df1.unpersist
- 将df5或df4重新分区为16、64、192、256、1000、4000(尽管数据看起来不倾斜)
- 将spark.sql.shuffle.partitions更改为16,64,192,256,1000,4000
- 每个执行器使用1个和2个内核,以便每个任务都有更多内存。
- 具有2个执行器和2个核心或4个核心。
- 将“spark.memory.fraction”更改为0.8,将“spark.memory.storagefraction”更改为0.4.
总是同样的错误!怎么可能把这些记忆都吹走??有没有可能df实际上不适合内存?如果您需要任何其他信息或打印屏幕,请让我知道。
编辑1
我将集群更改为2个spark workers和1个executor,每个executor的spark.sql.shuffle.partitions=48。每个执行器有115G和8个核心。下面是我加载文件(2.2GB)的代码,将每一行转换为密集向量,并向PCA提供信息。
文件中的每一行都具有以下格式(4.568行,每行40.107个双值):
"[x1,x2,x3,...]"
和代码:
Dataset<Row> df1 = sp.read().format("com.databricks.spark.csv").option("header", "true").load("/home/ubuntu/yolo.csv");
StructType schema2 = new StructType(new StructField[] {
new StructField("intensity",new VectorUDT(),false,Metadata.empty())
});
Dataset<Row> df = df1.map((Row originalrow) -> {
String yoho =originalrow.get(0).toString();
int sizeyoho=yoho.length();
String yohi = yoho.substring(1, sizeyoho-1);
String[] yi = yohi.split(",");
int s = yi.length;
double[] tmplist= new double[s];
for(int i=0;i<s;i++){
tmplist[i]=Double.parseDouble(yi[i]);
}
Row newrow = RowFactory.create(Vectors.dense(tmplist));
return newrow;
}, RowEncoder.apply(schema2));
PCAModel pcaexp = new PCA()
.setInputCol("intensity")
.setOutputCol("pcaFeatures")
.setK(2)
.fit(df);
ERROR Executor: Exception in task 1.0 in stage 6.0 (TID 43)
java.lang.OutOfMemoryError
at java.io.ByteArrayOutputStream.hugeCapacity(ByteArrayOutputStream.java:123)
at java.io.ByteArrayOutputStream.grow(ByteArrayOutputStream.java:117)
at java.io.ByteArrayOutputStream.ensureCapacity(ByteArrayOutputStream.java:93)
at java.io.ByteArrayOutputStream.write(ByteArrayOutputStream.java:153)
at org.apache.spark.util.ByteBufferOutputStream.write(ByteBufferOutputStream.scala:41)
at java.io.ObjectOutputStream$BlockDataOutputStream.drain(ObjectOutputStream.java:1877)
at java.io.ObjectOutputStream$BlockDataOutputStream.setBlockDataMode(ObjectOutputStream.java:1786)
at java.io.ObjectOutputStream.writeObject0(ObjectOutputStream.java:1189)
at java.io.ObjectOutputStream.writeObject(ObjectOutputStream.java:348)
at org.apache.spark.serializer.JavaSerializationStream.writeObject(JavaSerializer.scala:43)
at org.apache.spark.serializer.JavaSerializerInstance.serialize(JavaSerializer.scala:100)
at org.apache.spark.executor.Executor$TaskRunner.run(Executor.scala:456)
at java.util.concurrent.ThreadPoolExecutor.runWorker(ThreadPoolExecutor.java:1149)
at java.util.concurrent.ThreadPoolExecutor$Worker.run(ThreadPoolExecutor.java:624)
at java.lang.Thread.run(Thread.java:748)
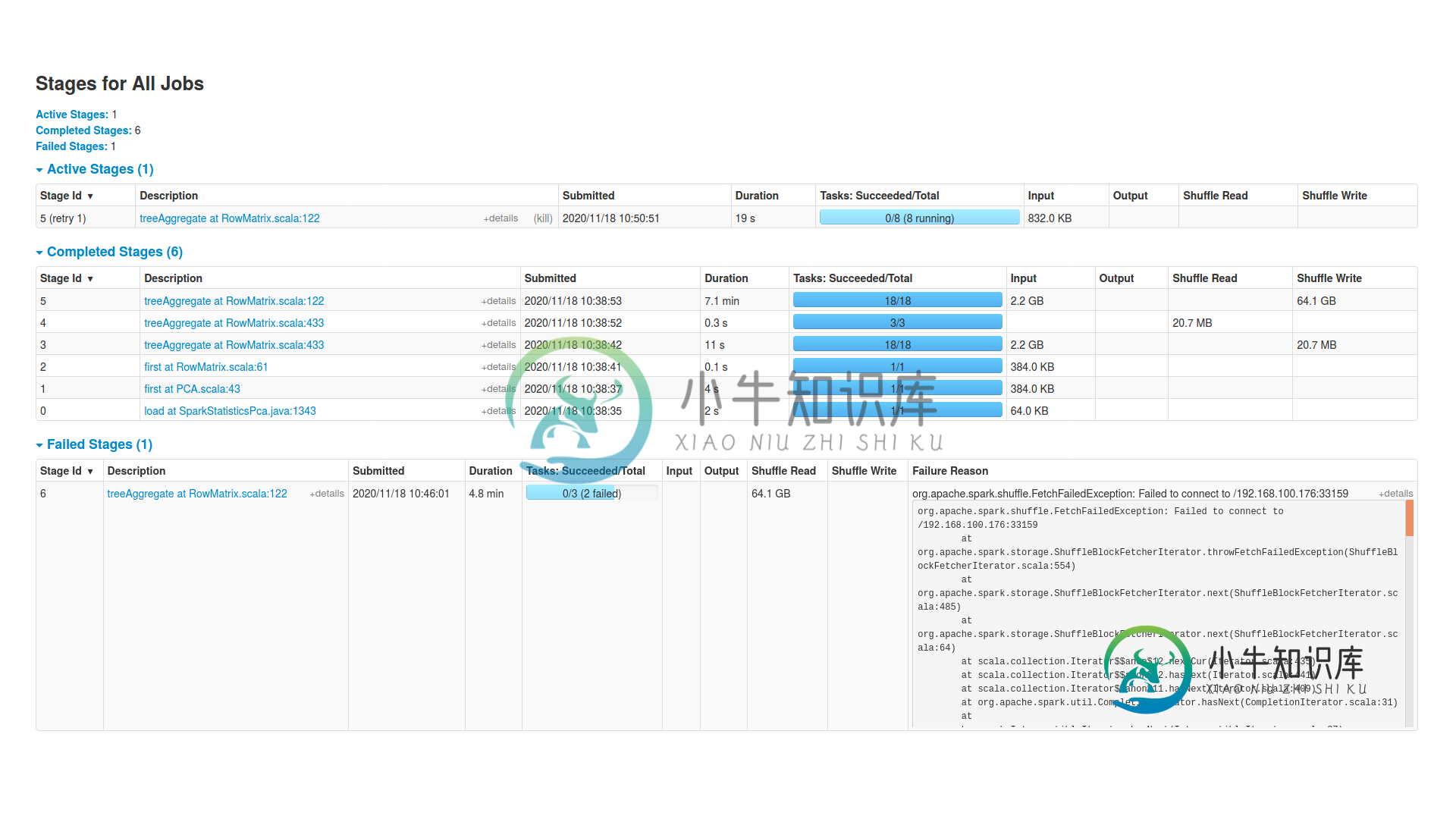
这是失败的阶段(rowmatrix.scala:122):
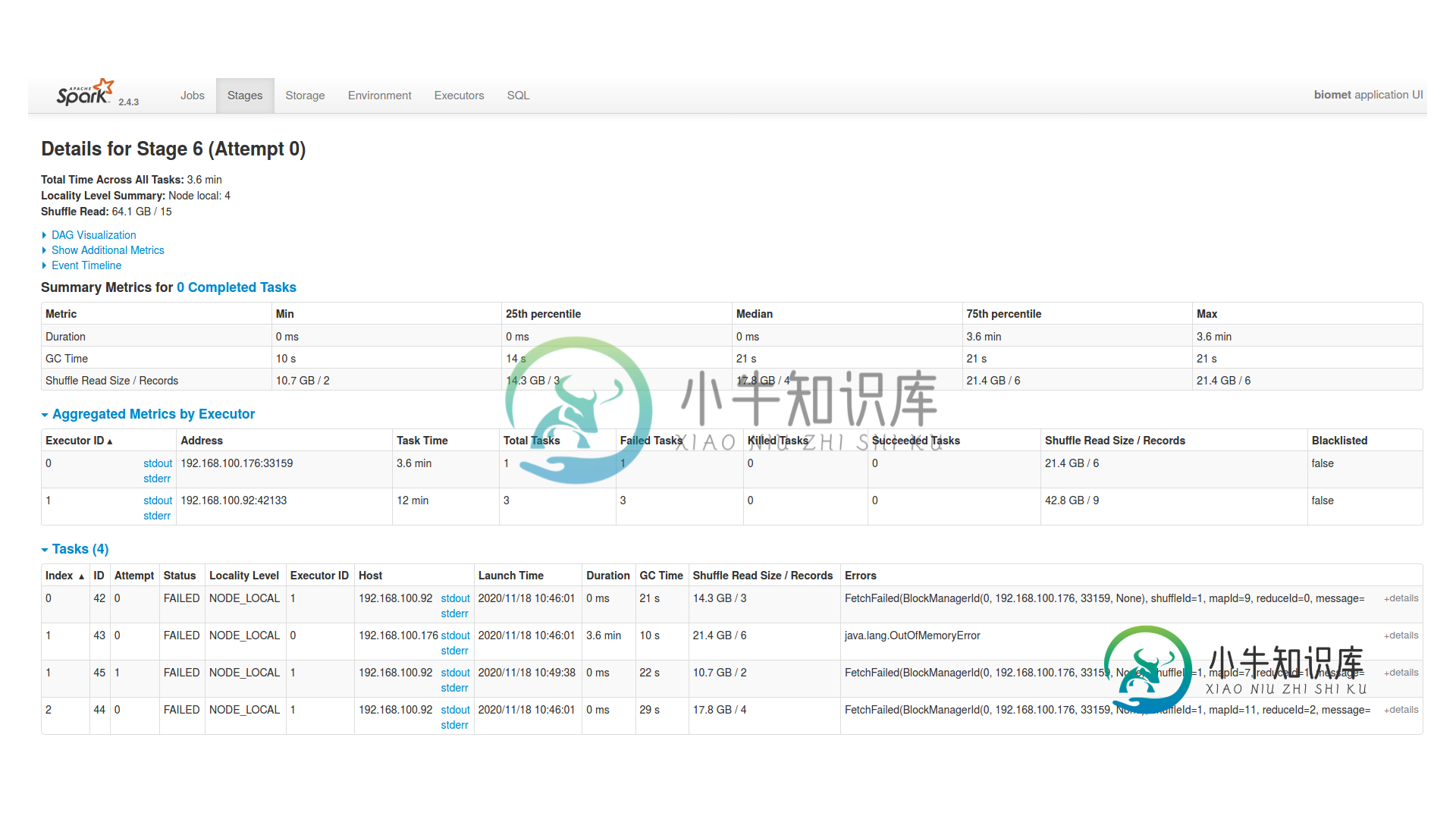
编辑2

编辑3
Dataset<Row> df1 = sp.read().format("com.databricks.spark.csv").option("header", "true").load("/home/ubuntu/yolo.csv");
StructType schema2 = new StructType(new StructField[] {
new StructField("intensity",new VectorUDT(),false,Metadata.empty())
});
Dataset<Row> df = df1.map((Row originalrow) -> {
String yoho =originalrow.get(0).toString();
int sizeyoho=yoho.length();
String yohi = yoho.substring(1, sizeyoho-1);
String[] yi = yohi.split(",");//this string array has all 40.107 values
int s = yi.length;
double[] tmplist= new double[s];
for(int i=0;i<10;i++){//I narrow it down to take only the first 10 values of each row
tmplist[i]=Double.parseDouble(yi[i]);
}
Row newrow = RowFactory.create(Vectors.dense(tmplist));
return newrow;
}, RowEncoder.apply(schema2));
PCAModel pcaexp = new PCA()
.setInputCol("intensity")
.setOutputCol("pcaFeatures")
.setK(2)
.fit(df);
共有1个答案
当Spark应用程序进行大的洗牌阶段,试图在执行器之间重新分配大量数据,并且集群网络中存在一些问题时,就会出现“丢失洗牌的输出位置”。
Spark说你在某些阶段没有记忆。您正在执行需要不同阶段的转换,它们也消耗内存。此外,您首先要持久化dataframe,并且应该检查存储级别,因为在内存中持久化是肯定的。
您正在链接几个Spark范围内的转换:例如,执行第一个pivot stage时,Spark创建了一个stage并为您的列执行shuffle分组,可能您有数据倾斜,并且执行器消耗的内存比其他执行器多得多,可能错误可能发生在其中一个执行器中。
PCA估计器除了对数据树进行变换外,还将数据树转换为RDD,增加了计算协方差矩阵的内存,并对NxN元素的微风矩阵进行密集表示。例如,SVD是用微风制作的。这给其中一个执行者带来了很大的压力。
也许您可以在HDFS(或其他任何东西)中保存得到的数据帧,并将PCA作为另一个Spark应用程序。
主要的问题。在de SVD之前,算法需要计算语法矩阵,它使用RDD中的树聚合。这将创建一个非常大的双矩阵,将被发送到驱动程序,并有错误,因为您的驱动程序没有足够的内存。您需要大幅增加驱动程序内存。你有网络错误,如果一个执行器失去连接,作业崩溃,它不会尝试重新执行。
就我个人而言,我会尝试在微风(或微笑)中直接在驱动程序中进行PCA,我的意思是,收集RDD字段,因为数据集比协方差矩阵小得多,并用浮动表示手动进行。
只使用Breeze来计算主成分分析的代码,而不使用Spark或Treeagregation:
import breeze.linalg._
import breeze.linalg.svd._
object PCACode {
def mean(v: Vector[Double]): Double = v.valuesIterator.sum / v.size
def zeroMean(m: DenseMatrix[Double]): DenseMatrix[Double] = {
val copy = m.copy
for (c <- 0 until m.cols) {
val col = copy(::, c)
val colMean = mean(col)
col -= colMean
}
copy
}
def pca(data: DenseMatrix[Double], components: Int): DenseMatrix[Double] = {
val d = zeroMean(data)
val SVD(_, _, v) = svd(d.t)
val model = v(0 until components, ::)
val filter = model.t * model
filter * d
}
def main(args: Array[String]) : Unit = {
val df : DataFrame = ???
/** Collect the data and do the processing. Convert string to double, etc **/
val data: Array[mutable.WrappedArray[Double]] =
df.rdd.map(row => (row.getAs[mutable.WrappedArray[Double]](0))).collect()
/** Once you have the Array, create the matrix and do the PCA **/
val matrix = DenseMatrix(data.toSeq:_*)
val pcaRes = pca(matrix, 2)
println("result pca \n" + pcaRes)
}
}
-
我试图在Eclipse和GGTS上编译我的Grails项目,在这两个平台上每次都收到相同的错误: 我已经激活了堆空间查看器,但它从来没有达到我在eclipse.ini和ggts.ini上定义的1GB最大大小(甚至没有达到400M),所以我想问题是我的MAC上的JVM选项应该改变。 我设置的Eclipse和GGTS值: -xx:permsize=1024m java-xx:+printflagsfi
-
我面临一些关于内存问题的问题,但我无法解决它。非常感谢您的帮助。我不熟悉Spark和pyspark功能,试图读取大约5GB大小的大型JSON文件,并使用 每次运行上述语句时,都会出现以下错误: 我需要以RDD的形式获取JSON数据,然后使用SQLSpark进行操作和分析。但是我在第一步(读取JSON)本身就出错了。我知道要读取如此大的文件,需要对Spark会话的配置进行必要的更改。我遵循了Apac
-
我正在尝试在本地机器上运行数据处理管道的拷贝,它可以在集群上正常工作,并且hadoop和hbase可以在独立模式下工作。Pipeline包含几个相继启动的mapreduce作业,其中一个作业有mapper,它不在输出中写入任何内容(依赖于输入,但在我的测试中它不写入任何内容),但有Reducer。在此作业运行期间收到此异常: 我检查了mapper生成的文件,我预计它们将是空的,因为mapper没有
-
本文向大家介绍java和js实现的洗牌小程序,包括了java和js实现的洗牌小程序的使用技巧和注意事项,需要的朋友参考一下 这几天刚学了java和javascript,简单写了个用java和javascript的小程序 JavaScript的 java的 总结 以上所述是小编给大家介绍的java和js实现的洗牌小程序,希望对大家有所帮助,如果大家有任何疑问请给我留言,小编会及时回复大家的。在此也非
-
问题内容: 这是声明Java数组的常用方法: 但是此数组正在使用堆空间。有没有一种方法可以使用像c ++这样的堆栈空间来声明数组? 问题答案: 一言以蔽之。 存储在堆栈中的唯一变量是基元和对象引用。在您的示例中,引用存储在堆栈上,但它引用堆上的数据。 如果由于要确保清理内存而问来自C ++的问题,请阅读有关垃圾回收的信息。简而言之,Java自动处理清理堆中的内存以及堆栈中的内存。

