dplyr根据是否在组中进行总结
我正在寻找一种方法来制作一个汇总表,将R中某个因子的每个级别的值与该因子的所有其他级别的值进行比较。虹膜数据集的一个例子是,我想将setosa与所有其他人(即,versicolor和virginica)进行比较,然后将versicolor与其他人(setosa和virginica)进行比较,最后将virignica与其他人(versicolor和setosa)进行比较。在我的实际数据集中,我有许多组,所以我不想硬编码每个级别。如果可能的话,我正在寻找一个简单的解决方案。我想要的结果是一个如下所示的汇总表:
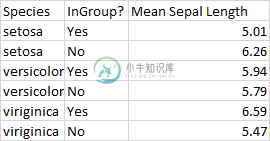
这里-组中的“是”是属于该组的物种(因此对于Setosa,它将仅是setosa),而“否”是不属于该组的物种(因此对于Setosa-不,它将是versicolor和virgnicia的组合)。
共有2个答案
1)我们可以在< code>dplyr本身中完成这项工作。通过使用< code>cur_group_id()(不使用除< code>dplyr之外的任何其他包)将列的< code >平均值和完整数据列的子集连接起来,按“物种”分组,< code >总结我们需要的列,即“Sepal.length”
library(dplyr)
iris %>%
group_by(Species) %>%
summarise(InGroup = c('Yes', 'No'), MeanSepalLength = c(mean(Sepal.Length),
mean(.$Sepal.Length[as.numeric(.$Species) != cur_group_id()])),
.groups = 'drop')
# A tibble: 6 x 3
# Species InGroup MeanSepalLength
# <fct> <chr> <dbl>
#1 setosa Yes 5.01
#2 setosa No 6.26
#3 versicolor Yes 5.94
#4 versicolor No 5.80
#5 virginica Yes 6.59
#6 virginica No 5.47
2)如果我们想在多列中这样做,请使用< code > cross
iris %>%
group_by(Species) %>%
summarise(InGroup = c('Yes', 'No'),
across(where(is.numeric), ~ c(mean(.),
mean(iris[[cur_column()]][
as.numeric(iris$Species) != cur_group_id()])), .names = 'Mean{.col}'),
.groups = 'drop')
-输出
# A tibble: 6 x 6
# Species InGroup MeanSepal.Length MeanSepal.Width MeanPetal.Length MeanPetal.Width
# <fct> <chr> <dbl> <dbl> <dbl> <dbl>
#1 setosa Yes 5.01 3.43 1.46 0.246
#2 setosa No 6.26 2.87 4.91 1.68
#3 versicolor Yes 5.94 2.77 4.26 1.33
#4 versicolor No 5.80 3.20 3.51 1.14
#5 virginica Yes 6.59 2.97 5.55 2.03
#6 virginica No 5.47 3.10 2.86 0.786
3)如果我们需要一个函数,也可以创建
f1 <- function(dat, grp) {
grp_str <- rlang::as_string(rlang::ensym(grp))
dat %>%
group_by({{grp}}) %>%
summarise(InGroup = c('Yes', 'No'),
across(where(is.numeric), ~ c(mean(.),
mean(dat[[cur_column()]][
as.numeric(dat[[grp_str]]) != cur_group_id()])),
.names = 'Mean{.col}'), .groups = 'drop')
}
-测试
f1(iris, Species)
# A tibble: 6 x 6
# Species InGroup MeanSepal.Length MeanSepal.Width MeanPetal.Length MeanPetal.Width
# <fct> <chr> <dbl> <dbl> <dbl> <dbl>
#1 setosa Yes 5.01 3.43 1.46 0.246
#2 setosa No 6.26 2.87 4.91 1.68
#3 versicolor Yes 5.94 2.77 4.26 1.33
#4 versicolor No 5.80 3.20 3.51 1.14
#5 virginica Yes 6.59 2.97 5.55 2.03
#6 virginica No 5.47 3.10 2.86 0.786
或使用菱形
f1(diamonds, cut)
# A tibble: 10 x 9
# cut InGroup Meancarat Meandepth Meantable Meanprice Meanx Meany Meanz
# <ord> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
# 1 Fair Yes 1.05 64.0 59.1 4359. 6.25 6.18 3.98
# 2 Fair No 0.790 61.7 57.4 3920. 5.72 5.72 3.53
# 3 Good Yes 0.849 62.4 58.7 3929. 5.84 5.85 3.64
# 4 Good No 0.793 61.7 57.3 3933. 5.72 5.72 3.53
# 5 Very Good Yes 0.806 61.8 58.0 3982. 5.74 5.77 3.56
# 6 Very Good No 0.796 61.7 57.3 3919. 5.73 5.72 3.53
# 7 Premium Yes 0.892 61.3 58.7 4584. 5.97 5.94 3.65
# 8 Premium No 0.766 61.9 57.0 3709. 5.65 5.66 3.50
# 9 Ideal Yes 0.703 61.7 56.0 3458. 5.51 5.52 3.40
#10 Ideal No 0.861 61.8 58.5 4249. 5.88 5.88 3.63
4)或者另一种选择是取sum中的差异并除以行数的差异
iris %>%
group_by(Species) %>%
summarise(InGroup = c('Yes', 'No'), across(where(is.numeric),
~ c(mean(.), (sum(iris[[cur_column()]]) -
sum(.))/(nrow(iris) - n())), .names = 'Mean{.col}'), .groups = 'drop')
在下面的代码中,我们使用< code>map分别对每个级别的< code>Species进行操作。对于每次迭代,我们创建一个分组列< code>in.group,标记一行是否是给定种类的成员。然后,我们按组返回所有数字列的平均值:
library(tidyverse)
unique(as.character(iris$Species)) %>%
set_names() %>%
map_df(
~iris %>%
group_by(in.group = Species==.x) %>%
summarise(across(where(is.numeric), mean, .names="mean_{col}")),
.id="Species"
)
#> # A tibble: 6 x 6
#> Species in.group mean_Sepal.Length mean_Sepal.Width mean_Petal.Length
#> <chr> <lgl> <dbl> <dbl> <dbl>
#> 1 setosa FALSE 6.26 2.87 4.91
#> 2 setosa TRUE 5.01 3.43 1.46
#> 3 versicolor FALSE 5.80 3.20 3.51
#> 4 versicolor TRUE 5.94 2.77 4.26
#> 5 virginica FALSE 5.47 3.10 2.86
#> 6 virginica TRUE 6.59 2.97 5.55
#> # … with 1 more variable: mean_Petal.Width <dbl>
您还可以将以下内容添加到链中,以使输出更加经济:
mutate(Species = case_when(in.group ~ Species,
!in.group ~ paste("not", Species))) %>%
select(-in.group)
这给出了:
Species mean_Sepal.Length mean_Sepal.Width mean_Petal.Length mean_Petal.Width
1 not setosa 6.26 2.87 4.91 1.68
2 setosa 5.01 3.43 1.46 0.246
3 not versicolor 5.80 3.20 3.51 1.14
4 versicolor 5.94 2.77 4.26 1.33
5 not virginica 5.47 3.10 2.86 0.786
6 virginica 6.59 2.97 5.55 2.03
您可以将它打包成一个函数:
compare.groups = function(data, group) {
group = ensym(group)
# Get levels of group
x = data %>%
distinct(!!group) %>%
pull(!!group) %>%
as.character %>%
set_names()
# Map over each level
x %>%
map_df(
~ data %>%
group_by(in.group = !!group == .x) %>%
summarise(across(where(is.numeric), mean, .names="mean_{col}")),
.id=as_label(enquo(group))
) %>%
mutate(!!group := case_when(in.group ~ !!group,
!in.group ~ paste("not", !!group))) %>%
select(-in.group)
}
# Run the function on a couple of data frames
compare.groups(iris, Species)
compare.groups(diamonds, cut)
您还可以使用该函数获取数据框中所有分类列的结果:
diamonds %>%
select(where(~!is.numeric(.))) %>%
names() %>%
set_names() %>%
map_df(
~compare.groups(diamonds, !!.x) %>%
rename(category = .x),
.id="variable"
)
variable category mean_carat mean_depth mean_table mean_price mean_x mean_y mean_z
1 cut not Ideal 0.861 61.8 58.5 4249. 5.88 5.88 3.63
2 cut Ideal 0.703 61.7 56.0 3458. 5.51 5.52 3.40
3 cut not Premium 0.766 61.9 57.0 3709. 5.65 5.66 3.50
4 cut Premium 0.892 61.3 58.7 4584. 5.97 5.94 3.65
5 cut not Good 0.793 61.7 57.3 3933. 5.72 5.72 3.53
6 cut Good 0.849 62.4 58.7 3929. 5.84 5.85 3.64
7 cut not Very Good 0.796 61.7 57.3 3919. 5.73 5.72 3.53
8 cut Very Good 0.806 61.8 58.0 3982. 5.74 5.77 3.56
9 cut not Fair 0.790 61.7 57.4 3920. 5.72 5.72 3.53
10 cut Fair 1.05 64.0 59.1 4359. 6.25 6.18 3.98
11 color not E 0.829 61.8 57.4 4123. 5.80 5.80 3.58
12 color E 0.658 61.7 57.5 3077. 5.41 5.42 3.34
13 color not I 0.772 61.7 57.4 3803. 5.68 5.68 3.50
14 color I 1.03 61.8 57.6 5092. 6.22 6.22 3.85
15 color not J 0.778 61.7 57.4 3856. 5.69 5.69 3.51
16 color J 1.16 61.9 57.8 5324. 6.52 6.52 4.03
17 color not H 0.777 61.7 57.4 3832. 5.69 5.69 3.51
18 color H 0.912 61.8 57.5 4487. 5.98 5.98 3.70
19 color not F 0.811 61.8 57.5 3977. 5.76 5.76 3.55
20 color F 0.737 61.7 57.4 3725. 5.61 5.62 3.46
# … with 20 more rows
-
问题内容: 我正在使用Oracle SQL,并且想对一些“喜欢”函数结果的不同行进行分组。举例说明: 假设我有一个表MESA,其中的一列是一个巨大的字符串。 我正在计算与特定模式匹配的行数: 因此,假设此查询的结果为: 水果..afsafafasfa … RED_APPLE 20 水果..afsafafasfa … YELLOW_APPLE 12 水果..afsafafasfa … GREEN_A
-
问题内容: 在MySQL中,我试图找到一种有效的方法来执行更新(如果表中已经存在一行),或者执行INSERT(如果该行不存在)。 到目前为止,我发现了两种可能的方法: 一个明显的例子:打开一个事务,选择以查找该行是否存在,如果不存在则进行INSERT,如果存在则进行UPDATE,提交事务 首先将IGNORE插入表中(如果该行已存在,则不会引发错误),然后进行更新 第二种方法避免了交易。 您认为哪一
-
我有两个整数数组,我试图根据另一个数组对第一个数组进行排序。 例如。和 b = {1,2,2,0,0, 在 B 中排序的值是 A 中每个整数的实值 排序后我期望的预期结果是: 这是我用的代码 它给出了我的输出:<code>a={2,3,1,0,0,6}和
-
我的数据框就像这个 由reprex包(v2.0.1)于2022-02-17创建 我想在grouping_bycol1之后根据count_col1和count_col2选择具有最大值的行。 我希望我的数据看起来像这样 你可以写一篇专栏文章 但不是两个人
-
假设我有以下Bus对象数组: 在总线数组填满之后,我想按总线号对总线数组进行排序(这是一个字符串,例如“501”)。每个总线对象都有一个总线号(总线[index].number)。没有重复的公共汽车号码。我怎样才能做到这一点?我看到了过滤器,但我不确定如何应用它。
-
问题内容: 我正在尝试优化使用MySQL 5.1中的视图的查询。似乎即使我从视图中选择1列,它始终会进行全表扫描。那是预期的行为吗? 该视图只是我在下面的第一个查询中指定的表的SELECT“这些表中的所有列-NOT *”。 这是当我从组成视图的查询中选择索引列PromotionID时的解释输出。如您所见,它与视图上的输出有很大的不同。 当我从视图中选择相同的东西时的输出 问题答案: MySQL中的

